降维
当我们处理待处理样本数据的特征过多,而其中有些特征有有些重复,计算起来速度较慢,我们可以采用降维算法。 假使我们要采用两种不的仪器来测量一些东西的尺寸,其中一个仪器测量结果的单位是英寸,另一个仪器测量的结果是厘米,我们希望将测量的结果作为我们机器学习的特征。现在的问题的是,两种仪器对同一个东西测量的结果不完全相等(由于误差、精度等),而将两者都作为特征有些重复,因而,我们希望将这个二维的数据降至一维.将数据从二维降至一维的过程就叫做降维。
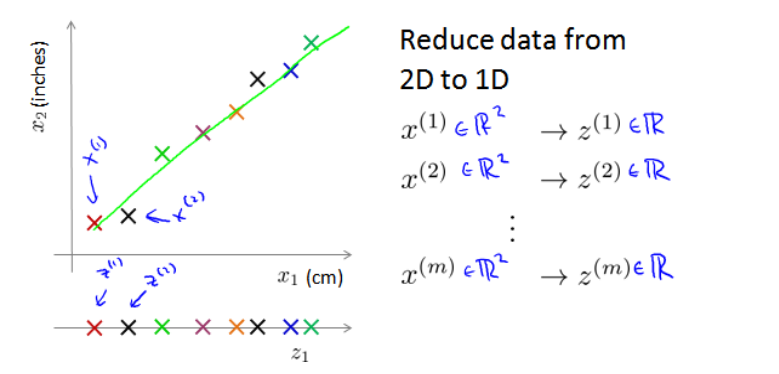
如图所示,将所有在一个平面上的样本点转换到一条直线上,将一组两维的数据用一组一维的来表示,这就是将二维数据降为一维。
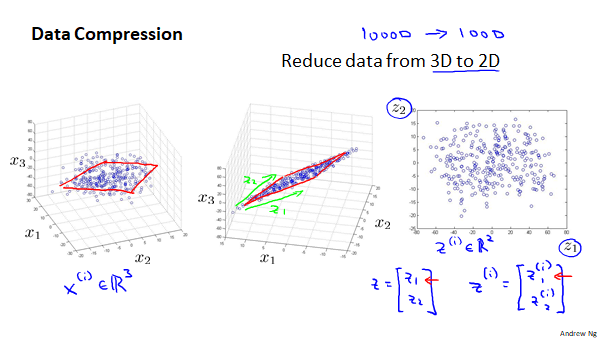
而对于三维的数据我们可以降维两维,将所有的样本点投影至一个平面。
为什么要降维
数据压缩
数据压缩可以使用较少的计算机内存或磁盘空间,同时也可以加速我们的学习算法。对于一些特征数量较高的样本数据,如果不进行数据压缩,计算量会非常的大,学习的速度太慢。
数据可视化
在许多机器学习问题中,如果我们能将数据可视化,我们就有可能寻找到一个更好的解决方案。这时候就用到了降维。
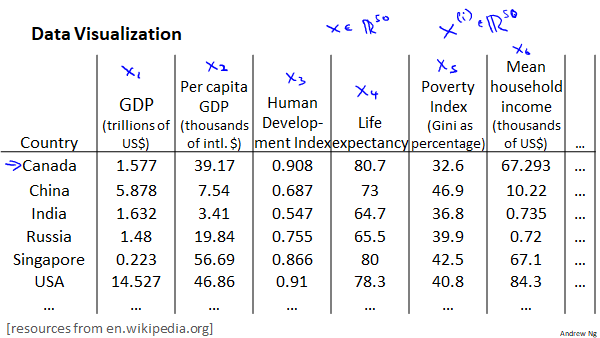
假设我们有许多关于不同国家的数据,每个特征向量都有50个特征,如果要将这50维数据可视化是不可能的的,如果我们能通过降维的方法将其降至2维,就可以进行可视化了。
主成成分分析(PCA)算法
主成成分分析(Principal components analysis 简称PCA)是最重要的的降维方法之一。。PCA算法要做的就是找出数据里面的主要的方面,用其来代替原始的数据。在PCA中,我们要做的就是找到一个方向向量,当把所有的数据都投射到该向量上时,我们希望投射的平均均方误差尽可能的小。
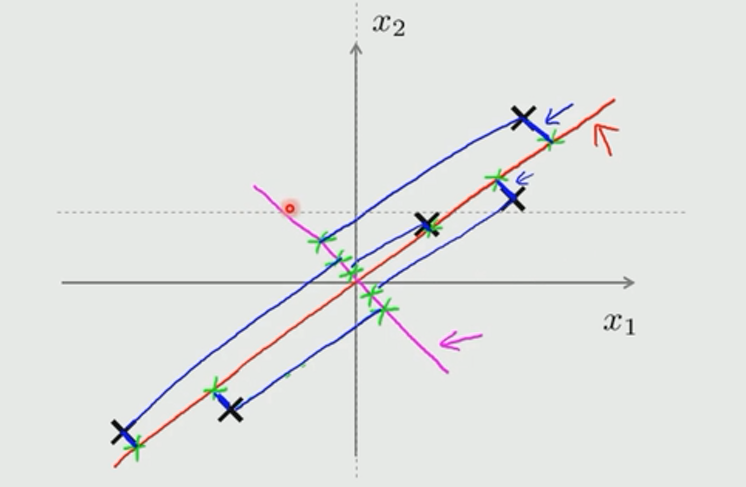
在图中所示的数据中,我们选择红色的线作为方向向量,而不选择粉红色的线就是应为其平均均方误差小。
PCA与线性回归的区别
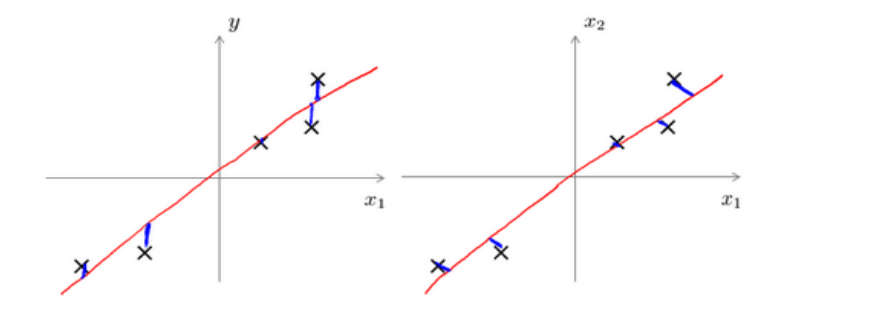
如图所示,左边是线性回归的误差(垂直于横轴方向),线性回归尝试最小化的是预测误差,来预测结果;右边是PCA的误差,垂直于方向向量,最小化的是投射误差,并不会做任何预测。
PCA算法流程
1.数据预处理:中心化X-x
2.求样本的协方差矩阵m1XXT
3.对协方差m1XXT矩阵做特征值分解。
4.选出最大的k个特征值对于的k个特征向量
5.将原始数据投影到选取的特征向量上。
6.输出投影后的数据集。
证明过程看不太懂😭,所以这里仅给出算法流程,具体证明过程可以去看下面这篇博客:主成分分析(PCA)原理总结
PCA实现
python实现
1
2
| import numpy as np
import matplotlib.pylab as plt
|
1
2
3
4
5
6
7
|
data=np.genfromtxt('data.csv',delimiter=',')
x_data=data[:,0]
y_data=data[:,1]
plt.scatter(x_data,y_data)
plt.show()
data.shape
|
(100, 2)
1
2
3
4
5
6
|
def zeroMean(dataMat):
meanMaT=np.mean(dataMat,axis=0)
newMat=dataMat-meanMaT
return newMat,meanMaT
|
1
2
3
4
| newMat,meanMat=zeroMean(data)
covMat=np.cov(newMat,rowvar=0)
covMat
|
array([[ 94.99190951, 125.62024804],
[125.62024804, 277.49520751]])
1
2
|
newMat.T.dot(newMat)/newMat.shape[0]
|
array([[ 94.04199041, 124.36404556],
[124.36404556, 274.72025544]])
1
2
|
eigVal,eigVects=np.linalg.eig(np.mat(covMat))
|
array([ 30.97826888, 341.50884814])
matrix([[-0.89098665, -0.45402951],
[ 0.45402951, -0.89098665]])
1
2
3
|
eigValIndex=np.argsort(eigVal)
eigValIndex
|
array([0, 1], dtype=int64)
1
2
3
4
| k=1
n_eigValIndex=eigValIndex[-1:-(k+1):-1]
n_eigValIndex
|
array([1], dtype=int64)
1
2
3
|
n_eigVect=eigVects[:,n_eigValIndex]
n_eigVect
|
matrix([[-0.45402951],
[-0.89098665]])
1
2
3
|
lowdata=newMat*n_eigVect
lowdata[:5,:]
|
matrix([[ 44.02694787],
[ 1.49722533],
[ 3.35564513],
[ 1.73205523],
[-17.84406034]])
1
2
3
|
recoMat=(lowdata*n_eigVect.T)+meanMat
recoMat[:5,:]
|
matrix([[28.96880808, 33.50762783],
[48.27855698, 71.40104277],
[47.43477956, 69.74521555],
[48.17193728, 71.19181247],
[57.06007136, 88.63387007]])
1
2
3
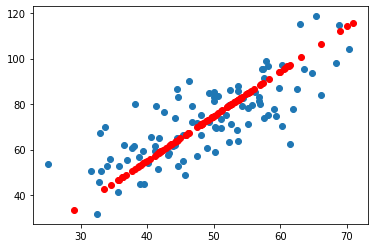
| plt.scatter(x_data,y_data)
plt.scatter(np.array(recoMat)[:,0],np.array(recoMat)[:,1],c='r')
plt.show()
|
sklearn实现
1
2
3
| from sklearn.decomposition import PCA
model=PCA(n_components=1)
model.fit(data)
|
PCA(n_components=1)
1
| model.explained_variance_
|
array([341.50884814])
1
| model.explained_variance_ratio_
|
array([[0.45402951, 0.89098665]])
1
2
| new_data=model.transform(data)
new_data[:5,:]
|
array([[-44.02694787],
[ -1.49722533],
[ -3.35564513],
[ -1.73205523],
[ 17.84406034]])
1
2
3
4
| recdata=model.inverse_transform(new_data)
plt.scatter(data[:,0],data[:,1])
plt.scatter(recdata[:,0],recdata[:,1],c='r')
plt.show()
|
PCA算法优缺点
优点
1.仅仅需要以方差衡量信息量,不受数据集以外的因素影响
2.各主成成分之间正交,可以消除原始数据成分间的相互影响的因素
3.计算方法简单,主要运算是特征值分解,易于实现
缺点
1.主成成分各个特征维度的含义具有一定的模糊性,不如原始样本解释性强
2.方差小的非主成成分也可能含有对样本差异的重要信息,因降维丢弃可能对后续数据处理有影响









